2022年7月,beat365正版唯一吳小惠教授團隊在生物信息學知名期刊《Briefings in Bioinformatics》(最新影響因子11.622)發表題為“PolyAtailor: measuring poly(A) tail length from short-read and long-read sequencing data”的研究論文。


真核生物基因表達過程中,剪切(Cleavage)與多聚腺苷化[Polyadenylation或Poly(A)]是形成mRNA所必需的加工步驟。Poly(A)尾巴長度的變化影響mRNA的核輸出、mRNA穩定性和翻譯,對基因表達起重要調控作用。吳小惠團隊建立了一個普适且高效的生物信息學分析框架—PolyTailor,用于識别和分析基于三代PacBio或二代短讀段測序的poly(A)尾巴。PolyAtailor提供了兩個核心功能--Tail_map和Tail_scan,對有或沒有參考基因組的物種均可進行分析,可為用戶提供詳細的poly(A)尾巴信息,包括尾巴位置、尾巴長度、尾巴序列和尾巴類型。此外,PolyTailor集成豐富的poly(A)尾巴和位點分析功能,如差異尾長分析、poly(A)位點識别和注釋,以及尾巴中堿基組成的統計和可視化等。通過使用來自HeLa樣本和拟南芥的三種測序數據,與另外三種方法--FLAMAnalysis、FLEPSeq和PAIsoSeq的比較結果表明,PolyAtailor具有更高的靈敏度和準确性。PolyAtailor程序可從https://github.com/BMILAB/PolyAtailor獲得。
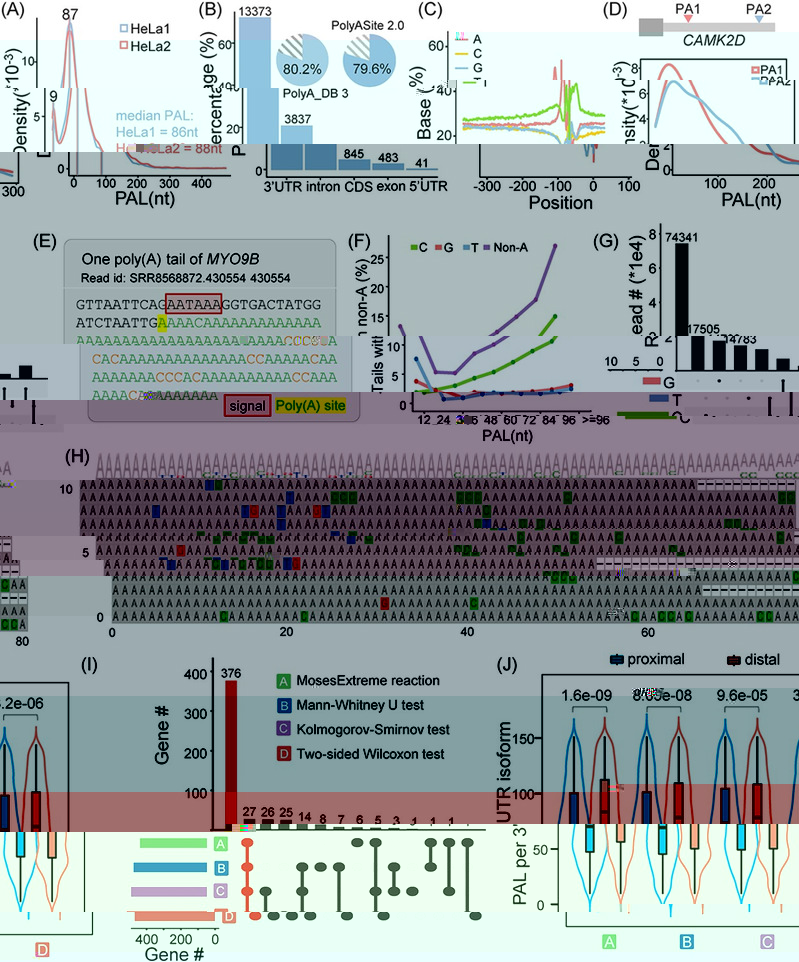
PolyAtailor可用于poly(A)尾巴動态的分析及可視化
beat365正版唯一為論文第一單位,beat365正版唯一吳小惠教授為論文唯一通訊作者,吳小惠團隊的碩士生劉夢飛為論文第一作者,碩士研究生郝琳琳和本科生楊思恩同學也參與此工作。該研究得到了國家自然科學基金面上項目支持。吳小惠教授長期緻力于生物和健康醫療大數據研究,特别是APA相關分析,團隊已經開發了多個生物信息學分析工具或數據庫,如scAPAdb、PlantAPAdb、APAtrap、scAPAtrap、movAPA等,成果發表于PNAS、Genome Research、Nucleic Acids Research、Briefings in Bioinformatics、Bioinformatics等期刊。
論文鍊接:
https://academic.oup.com/bib/article/23/4/bbac271/6620877
